AutoMLのPyCaretを使って、教師ありな分類を行う。
ライブラリインストール
!pip install numba==0.53 # 依存関係エラー対応
# https://stackoverflow.com/questions/70148065/numba-needs-numpy-1-20-or-less-for-shapley-import
!pip install pandas-profiling==3.1.0 # エラー対応 実行後にランタイム再起動
!pip install pycaret
from pycaret.utils import enable_colab
enable_colab()まずはColab用にインストール。2022.8現在は依存関係のエラーが出てしまうようなので、numba(処理スピードをアップさせるライブラリ)のバージョンを指定してインストールする。またpandas-profilingもバージョン指定する。実行後はランタイムを再起動する。
テストデータ読み込み
import pandas as pd
from sklearn.datasets import load_breast_cancer
load_data = load_breast_cancer()
tg_df_all = pd.DataFrame(load_data.data, columns = load_data.feature_names)
tg_df_all['y'] = load_data.target
tg_df_all
---
mean radius mean texture mean perimeter mean area mean smoothness mean compactness mean concavity mean concave points mean symmetry mean fractal dimension ... worst texture worst perimeter worst area worst smoothness worst compactness worst concavity worst concave points worst symmetry worst fractal dimension y
0 17.99 10.38 122.80 1001.0 0.11840 0.27760 0.30010 0.14710 0.2419 0.07871 ... 17.33 184.60 2019.0 0.16220 0.66560 0.7119 0.2654 0.4601 0.11890 0
1 20.57 17.77 132.90 1326.0 0.08474 0.07864 0.08690 0.07017 0.1812 0.05667 ... 23.41 158.80 1956.0 0.12380 0.18660 0.2416 0.1860 0.2750 0.08902 0
2 19.69 21.25 130.00 1203.0 0.10960 0.15990 0.19740 0.12790 0.2069 0.05999 ... 25.53 152.50 1709.0 0.14440 0.42450 0.4504 0.2430 0.3613 0.08758 0
3 11.42 20.38 77.58 386.1 0.14250 0.28390 0.24140 0.10520 0.2597 0.09744 ... 26.50 98.87 567.7 0.20980 0.86630 0.6869 0.2575 0.6638 0.17300 0
4 20.29 14.34 135.10 1297.0 0.10030 0.13280 0.19800 0.10430 0.1809 0.05883 ... 16.67 152.20 1575.0 0.13740 0.20500 0.4000 0.1625 0.2364 0.07678 0
... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ...
564 21.56 22.39 142.00 1479.0 0.11100 0.11590 0.24390 0.13890 0.1726 0.05623 ... 26.40 166.10 2027.0 0.14100 0.21130 0.4107 0.2216 0.2060 0.07115 0
565 20.13 28.25 131.20 1261.0 0.09780 0.10340 0.14400 0.09791 0.1752 0.05533 ... 38.25 155.00 1731.0 0.11660 0.19220 0.3215 0.1628 0.2572 0.06637 0
566 16.60 28.08 108.30 858.1 0.08455 0.10230 0.09251 0.05302 0.1590 0.05648 ... 34.12 126.70 1124.0 0.11390 0.30940 0.3403 0.1418 0.2218 0.07820 0
567 20.60 29.33 140.10 1265.0 0.11780 0.27700 0.35140 0.15200 0.2397 0.07016 ... 39.42 184.60 1821.0 0.16500 0.86810 0.9387 0.2650 0.4087 0.12400 0
568 7.76 24.54 47.92 181.0 0.05263 0.04362 0.00000 0.00000 0.1587 0.05884 ... 30.37 59.16 268.6 0.08996 0.06444 0.0000 0.0000 0.2871 0.07039 1sklearnの肺ガンデータを使って実験する。肺ガンがポジティブ(陽性)の場合、yが1をとる。
前処理
# 未見データの取り分け
tg_df = tg_df_all.sample(frac=0.90, random_state = 0).reset_index(drop = True)
tg_df_unseen = tg_df_all.drop(tg_df.index).reset_index(drop = True)
print('All data: ' + str(tg_df.shape))
print('Data for Modeling: ' + str(tg_df.shape))
print('Unseen Data for Predictions: ' + str(tg_df_unseen.shape))
-----
All data: (569, 31)
Data for Modeling: (512, 31)
Unseen Data for Predictions: (57, 31)まずは未見データを切り分ける。今回は9割を訓練用(512行)、1割を未見用(57行)とする。
前処理
# 分類を読み込み
from pycaret.classification import *
# 前処理
ret = setup(data = tg_df,
target = 'y', # 分類のターゲット
session_id = 0,
normalize = False,
train_size = 0.6, # train/testの分割数指定
silent = True) # 型推定の確認をスキップ# 分類方法の一覧
models()
-----
Name Reference Turbo
ID
lr Logistic Regression sklearn.linear_model._logistic.LogisticRegression True
knn K Neighbors Classifier sklearn.neighbors._classification.KNeighborsCl... True
nb Naive Bayes sklearn.naive_bayes.GaussianNB True
dt Decision Tree Classifier sklearn.tree._classes.DecisionTreeClassifier True
svm SVM - Linear Kernel sklearn.linear_model._stochastic_gradient.SGDC... True
rbfsvm SVM - Radial Kernel sklearn.svm._classes.SVC False
gpc Gaussian Process Classifier sklearn.gaussian_process._gpc.GaussianProcessC... False
mlp MLP Classifier sklearn.neural_network._multilayer_perceptron.... False
ridge Ridge Classifier sklearn.linear_model._ridge.RidgeClassifier True
rf Random Forest Classifier sklearn.ensemble._forest.RandomForestClassifier True
qda Quadratic Discriminant Analysis sklearn.discriminant_analysis.QuadraticDiscrim... True
ada Ada Boost Classifier sklearn.ensemble._weight_boosting.AdaBoostClas... True
gbc Gradient Boosting Classifier sklearn.ensemble._gb.GradientBoostingClassifier True
lda Linear Discriminant Analysis sklearn.discriminant_analysis.LinearDiscrimina... True
et Extra Trees Classifier sklearn.ensemble._forest.ExtraTreesClassifier True
lightgbm Light Gradient Boosting Machine lightgbm.sklearn.LGBMClassifier True
dummy Dummy Classifier sklearn.dummy.DummyClassifier True分類の学習方法一覧が出力される。ざっとこれくらいあるのかと把握したところで、実際に比較してみる。
# F1の値をソート軸に比較する
compare_models(sort = "F1", fold = 10)| index | Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | TT (Sec) |
|---|---|---|---|---|---|---|---|---|---|
| et | Extra Trees Classifier | 0.9673 | 0.9946 | 0.985 | 0.9669 | 0.9754 | 0.9265 | 0.9291 | 0.412 |
| ridge | Ridge Classifier | 0.9575 | 0.0 | 0.99 | 0.9495 | 0.9686 | 0.9027 | 0.9079 | 0.02 |
| dt | Decision Tree Classifier | 0.9575 | 0.9518 | 0.97 | 0.9659 | 0.9675 | 0.906 | 0.9076 | 0.022 |
| lightgbm | Light Gradient Boosting Machine | 0.9542 | 0.9895 | 0.98 | 0.9525 | 0.9656 | 0.8968 | 0.8997 | 0.109 |
| rf | Random Forest Classifier | 0.9542 | 0.9918 | 0.975 | 0.9571 | 0.9655 | 0.8972 | 0.8997 | 0.584 |
| qda | Quadratic Discriminant Analysis | 0.9545 | 0.9922 | 0.965 | 0.9657 | 0.9649 | 0.9001 | 0.902 | 0.021 |
| lda | Linear Discriminant Analysis | 0.9512 | 0.9893 | 0.98 | 0.9487 | 0.9635 | 0.8897 | 0.8932 | 0.027 |
| ada | Ada Boost Classifier | 0.9478 | 0.99 | 0.96 | 0.9611 | 0.96 | 0.8848 | 0.8872 | 0.165 |
| gbc | Gradient Boosting Classifier | 0.9411 | 0.9915 | 0.955 | 0.9572 | 0.9553 | 0.868 | 0.8714 | 0.335 |
| nb | Naive Bayes | 0.9413 | 0.9896 | 0.96 | 0.9524 | 0.955 | 0.8695 | 0.8741 | 0.023 |
| lr | Logistic Regression | 0.9346 | 0.9887 | 0.965 | 0.9385 | 0.9511 | 0.8524 | 0.8551 | 0.475 |
| knn | K Neighbors Classifier | 0.9317 | 0.9572 | 0.97 | 0.933 | 0.9495 | 0.8439 | 0.853 | 0.129 |
| svm | SVM – Linear Kernel | 0.9017 | 0.0 | 0.9 | 0.9547 | 0.917 | 0.7954 | 0.8128 | 0.022 |
| dummy | Dummy Classifier | 0.6516 | 0.5 | 1.0 | 0.6516 | 0.789 | 0.0 | 0.0 | 0.011 |
colabのmarkdown形式での表コピー機能めっちゃ便利や…。
モデル構築
爆速で計算できる、リッジ回帰を使ってみる。リッジ回帰は過学習を防ぐ、正則化が取り入れられた線形モデルの回帰。
訓練データよりテストデータへの精度が高くなることが目標なので、モデルの複雑化を防ぎ、訓練データにフィットし過ぎないように計らってくれる方法論。
ridge = create_model("ridge", fold = 10)| Fold | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC |
|---|---|---|---|---|---|---|---|
| 0 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 1.0 | 1.0 |
| 1 | 0.9355 | 0.0 | 1.0 | 0.9091 | 0.9524 | 0.8531 | 0.8624 |
| 2 | 0.9355 | 0.0 | 0.95 | 0.95 | 0.95 | 0.8591 | 0.8591 |
| 3 | 0.9677 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9281 | 0.9305 |
| 4 | 0.9677 | 0.0 | 0.95 | 1.0 | 0.9744 | 0.931 | 0.9332 |
| 5 | 0.9355 | 0.0 | 1.0 | 0.9091 | 0.9524 | 0.8531 | 0.8624 |
| 6 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 1.0 | 1.0 |
| 7 | 0.9 | 0.0 | 1.0 | 0.8696 | 0.9302 | 0.7568 | 0.7802 |
| 8 | 0.9667 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9231 | 0.9258 |
| 9 | 0.9667 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9231 | 0.9258 |
| Mean | 0.9575 | 0.0 | 0.99 | 0.9495 | 0.9686 | 0.9027 | 0.9079 |
| Std | 0.0296 | 0.0 | 0.02 | 0.0416 | 0.0212 | 0.0702 | 0.0645 |
チューニングする。
# ridgeをチューニング
tuned_ridge = tune_model(ridge,
optimize = 'F1',
fold = 10,
n_iter = 100)
tuned_ridge| Fold | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC |
|---|---|---|---|---|---|---|---|
| 0 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 1.0 | 1.0 |
| 1 | 0.9355 | 0.0 | 1.0 | 0.9091 | 0.9524 | 0.8531 | 0.8624 |
| 2 | 0.9355 | 0.0 | 0.95 | 0.95 | 0.95 | 0.8591 | 0.8591 |
| 3 | 0.9677 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9281 | 0.9305 |
| 4 | 0.9677 | 0.0 | 0.95 | 1.0 | 0.9744 | 0.931 | 0.9332 |
| 5 | 0.9677 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9281 | 0.9305 |
| 6 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 1.0 | 1.0 |
| 7 | 0.9 | 0.0 | 1.0 | 0.8696 | 0.9302 | 0.7568 | 0.7802 |
| 8 | 0.9667 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9231 | 0.9258 |
| 9 | 0.9667 | 0.0 | 1.0 | 0.9524 | 0.9756 | 0.9231 | 0.9258 |
| Mean | 0.9608 | 0.0 | 0.99 | 0.9538 | 0.9709 | 0.9102 | 0.9148 |
| Std | 0.0287 | 0.0 | 0.02 | 0.0394 | 0.0206 | 0.0685 | 0.0629 |
0.96 -> 0.97に改善。
# 分類モデルの評価
evaluate_model(tuned_ridge)
# ナビゲーションをクリックで評価を確認できるFeature Importance
# 特徴量の影響を確認する
plot_model(tuned_ridge, "feature")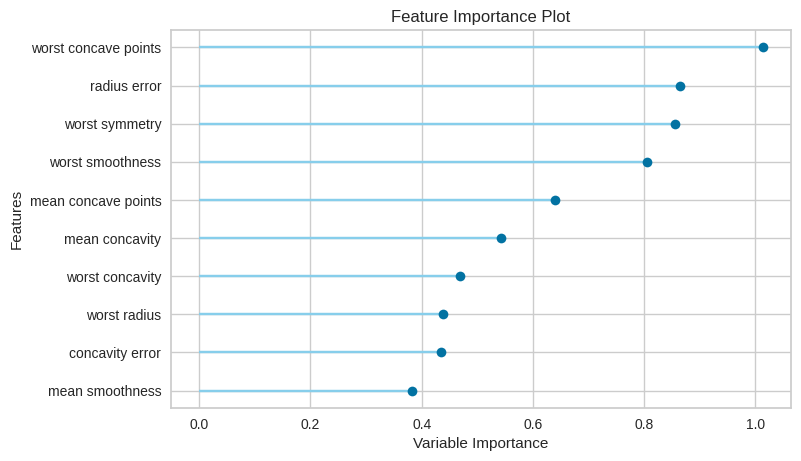
前回の回帰モデルと同様に、特徴量がそれぞれどの程度影響をもたらしているかを視覚的に確認できる。
混同マトリクス
# 混同マトリクス
plot_model(tuned_ridge, plot = "confusion_matrix")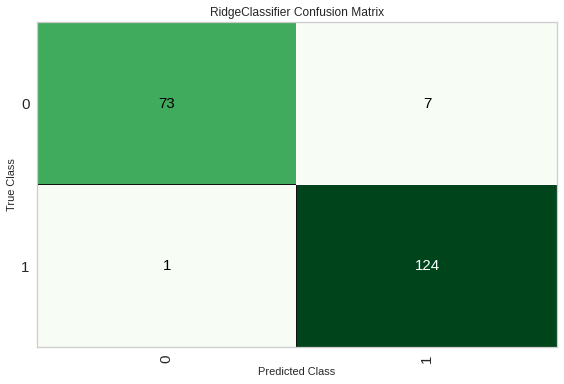
混同行列(confusion matrix)は、実際の値と予測した値の組み合わせを表で表現したもの。
True Positive -> 予測が陽性かつ真(正解)
True Negative -> 予測が陰性かつ真(正解)
False Positive -> 予測が陽性かつ偽(ハズレ)
False Negative -> 予測が陰性かつ偽(ハズレ)
この4パターンについて、どれだけ結果が配置されるかを確認することができる。
このマトリクスの場合、左上がTrue Negativeで右下がTrue Positiveとなり、高い精度で分類できていることが分かる。
https://note.nkmk.me/python-sklearn-confusion-matrix-score/
plot_model(tuned_ridge, "error")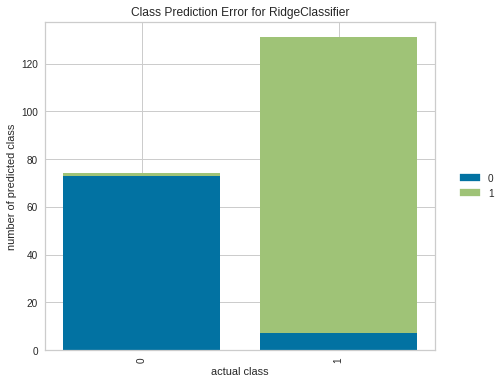
棒グラフで確認することもできる。X軸が実際のクラスのラベルで、それぞれ0と1で結果が棒グラフの形になる。
この形式でも比較的きれいに分類できていることが分かる。
モデルの確定
# モデルの確定
final_ridge = finalize_model(tuned_ridge)
# 最終モデルを使った予測
predict_model(final_ridge)
# 未見データに対して予測
predictions = predict_model(final_ridge, data = tg_df_unseen)
print(predictions)